COVIDready – Dezentrales SARS-CoV-2 Monitoring im Abwasser: Entwicklung einer validierten Analysemethode für abwassertechnische Labore auf Kläranlagen
Die EU-Kommission empfiehlt systematisch das Monitoring von SARS-CoV-2 im Abwasser zur Pandemiebekämpfung zu nutzen. Auch Deutschland ist dabei, ein nationales Monitoringsystem für SARS-CoV-2 und seine Virus-Varianten (Variants of Concern) an Pilotstandorten mit einer Probenahmefrequenz von zwei Abwasserproben pro Woche aufzubauen und die Analysedaten innerhalb von 48 Stunden den zuständigen Gesundheitsämtern zu melden.
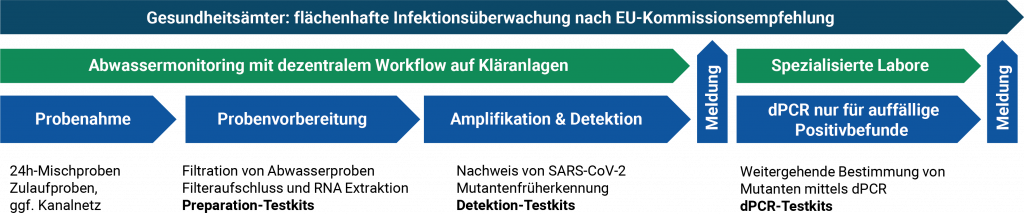
Für eine flächenhafte Anwendung des Abwassermonitorings fehlen in der dezentral organisierten deutschen Abwasserwirtschaft virologische Laborkapazitäten und etablierte Kommunikationswege mit dem öffentlichen Gesundheitsdienst. Alleine aus Zeitgründen ist ein Monitoring mit wenigen zentralisierten Laboren mit Probentransport, Aufbereitung, Analytik, Auswertung und Plausibilisierung der Daten in 48 Stunden kaum umsetzbar. Die Ausstattung von Kläranlagenlaboren mit molekularbiologischer quantitativer Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-qPCR)-Analytik würde erlauben, einen Großteil der Bevölkerung zu erfassen und gleichzeitig Analyseergebnisse mit Regionalkenntnis innerhalb der geforderten Zeit an die zuständigen Gesundheitsämter zu melden.
Dezentraler Workflow für abwassertechnische Labore
Die Zielsetzung des Verbundvorhabens COVIDready ist es, praxistaugliche Methoden zu optimieren und zu validieren, die es abwassertechnischen Laboren erlauben, mit vorgefertigten Testkits sowohl die Virenlast anhand der Virus-RNA zu quantifizieren wie auch als Frühwarnsystem für die Detektion ausgewählter Variants of Cornern zu dienen.
Das Verbundvorhaben wird vom Forschungsinstitut für Wasserwirtschaft und Klimazukunft an der RWTH Aachen e. V. koordiniert. Im Kooperationslabor von Emschergenossenschaft und Lippeverband (EGLV) wird ein teilautomatisierter Workflow für den dezentralen Einsatz in abwassertechnischen Laboren validiert und für den Praxistest ein Screening an drei Kläranlagen im EGLV-Verbandsgebiet durchgeführt. Ein Schulungsvideo wird für die Übertragung des Workflows auf andere Labore erstellt. Parallel wird im umweltanalytischen Labor des Instituts für Siedlungswasserwirtschaft (ISA) der RWTH Aachen der Workflow neu aufgebaut und hinsichtlich Sensitivität, Selektivität, Arbeitssicherheit, Handhabbarkeit, Schulungsbedarf, Verfügbarkeit und Kosten bewertet.
Der Nachweis von relevanten SARS-CoV-2 Variants of Concern im Abwasser ist neben zeitaufwändiger Gen-Sequenzierung wesentlich schneller und kostengünstiger mittels digitaler PCR (dPCR) möglich. Eine entsprechende Methode wird im Institut für Medizinische Virologie des Universitätsklinikums Frankfurt (KGU) etabliert und validiert. Die dPCR-Methode basiert hierbei auf einer Vereinzelung und Partitionierung der sich in der Probe befindlichen RNA-Molekülen, die in einer großen Anzahl separat ablaufender PCR-Reaktionen in getrennten Reaktionsräumen amplifiziert und nachgewiesen werden können. Vorteile dieser Methode sind eine hohe Amplifikationseffizienz, Unempfindlichkeit gegenüber PCR-Inhibitoren und eine niedrige Nachweisgrenze. Durch den Einsatz von mehreren Detektoren können auch Multiplex-Analysen durchgeführt werden, die zusätzlich zum reinen Nachweis von SARS-CoV-2-Genkopien eine Aussage zum Auftreten von Virusvarianten ermöglichen. Um die dynamische Ausbreitung kritischer Virusvarianten ohne teure und zeitintensive Sequenzierung überwachen zu können, wird ein Workflow vorgeschlagen, in dem ausgewählte und auffällige Proben mittels dPCR auf die Anwesenheit kennzeichnender Mutationen untersucht werden. Sensitivität und Spezifität dieser Methoden werden unter anderem durch „Spiken“ von SARS-CoV-2 negativen Abwasserproben mit authentischen, aber inaktiviertem Virusmaterial von SARS-CoV-2 Alpha, Beta oder Delta sowie anderen Coronaviren gesichert. Eine Validierung der Methodik wird durch internationale Ringversuche gewährleistet.
Zusammenarbeit zwischen Wasserwirtschaft und öffentlichem Gesundheitsdienst
Das Projektteam arbeitet mit dem öffentlichen Gesundheitsdienst im Untersuchungsgebiet zusammen, um dessen Anforderungen, Bedürfnisse und Erwartungen in Bezug auf eine umsetzbare Meldekette abzustimmen. Hierzu werden durch Lippeverband und FiW e. V. Vernetzungsworkshops mit Gesundheitsämtern, Wasserverbänden und Stadtentwässerungsbetrieben durchgeführt und der anvisierte Roll-out eines abwasserbasierten Monitorings untersucht und fachlich begleitet.
Das Verbundprojekt COVIDready trägt dazu bei, Grundlagern für die Einrichtung eines nationalen Monitoringsystems für das Coronavirus und seine Varianten zu legen. Darüber hinaus bieten die erforschten Lösungen Perspektiven für Biotechnologieunternehmen, mit ihren auf Abwasser abgestimmten molekularbiologischen Methoden und Analytiksystemen für SARS-CoV-2 neue Geschäftsfelder in der Abwasserepidemiologie aufzubauen. Mittelfristig können die Produkte auch zum Nachweis anderer Viren im Abwasser weiterentwickelt werden.
Verbundkoordinator
Dr. sc. Dipl.-Ing.
Frank-Andreas Weber
weber@fiw.rwth-aachen.de
Wissenschaftliche Leitung
Univ.-Prof. Dr.-Ing. habil.
Thomas Wintgens
wintgens@isa.rwth-aachen.de